2016年12月6日,清华大学生命学院、结构生物学高精尖创新中心陈柱成研究组在Nature发表题为“Structure and regulation of the chromatin remodeller ISWI”的研究论文。该工作通过X-ray晶体衍射的研究手段,解析了嗜热酵母的ISWI蛋白以及ISWI蛋白与组蛋白H4复合物的原子分辨率结构;结合相应的生化实验,揭示了ISWI蛋白的自抑制、被底物识别与激活以及感知接头DNA的长度,发挥染色质组装功能的分子机理。
染色质是真核生物的生命蓝图。染色质重塑蛋白利用ATP水解的能量,改变染色质结构,参与生命蓝图的绘制和重绘。ISWI是多个染色质重塑复合物的催化亚基。ISWI驱动核小体在基因组DNA上滑动,调控基因转录、异染色质形成、X-染色体失活以及其它重要的染色质活动。ISWI蛋白的催化核心(图a,Core)是一个自主的染色质重塑分子机器,其运作受严格的调控。ISWI活性受到AutoN结构域和NegC结构域的抑制作用,确保ISWI分子机器在没有结合底物时不会消耗ATP的能量。这些抑制作用分别被底物核小体的组蛋白H4尾巴和接头DNA拮抗。然而,组蛋白H4的乙酰化修饰削弱其对ISWI激活。这些多层次的调控作用确保细胞形成正确的高级染色质结构, 保证正常的生命活动。
该论文揭示了ISWI的AutoN包含两个抑制元件(图b, L3 和 a4),均与core2结合,使得ISWI处于抑制状态。组蛋白H4尾巴与core2的一个负电荷表面结合, 与其中一个AutoN抑制元件有竞争关系 (图c),从而解析了H4激活ISWI以及乙酰化作用细调ISWI活性的分子机理。另外,结合生化功能分析,该论文研究表明ISWI蛋白的NegC 与core2存在相互作用,这种相互作用是ISWI蛋白通过HSS结构域感知接头DNA长度、发挥染色质组装功能的分子基础 (图d-f)。
该论文是陈柱成研究组关于染色质重塑蛋白系列工作的一个重要组成部分。清华大学生命学院2014级直博生严丽娟和2016届硕士生王丽为论文的共同第一作者,陈柱成老师为通讯作者。该研究得到国家自然科学基金委、科技部、中组部、北京市结构生物学高精尖创新中心项目的经费支持,以及清华大学X-ray晶体平台和上海同步辐射光源中心的工作支持。
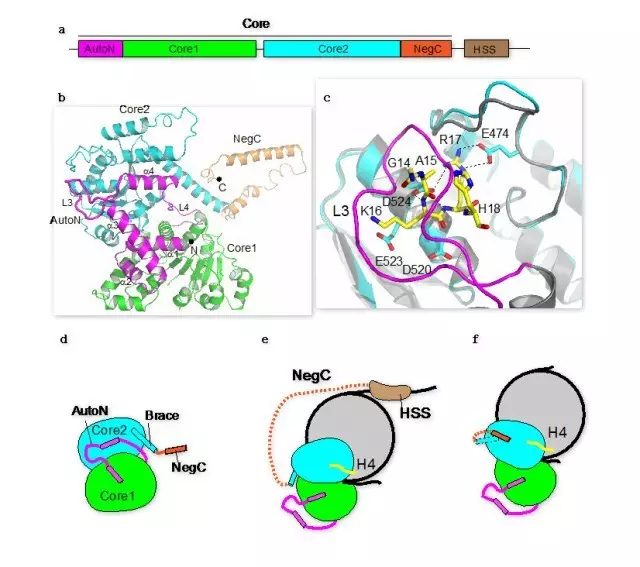
(a) Domain architecture of ISWI. (b) Overall structure of ISWI. (c) Superimposition of the structures of the core2 domain in the peptide-bound (cyan) and the auto-inhibited (grey) states. H4 peptide, yellow; L3 loop, magenta. (d) Cartoon image of ISWI in the auto-inhibited state. The tethering HSS domain is omitted. (e) Proposed model of ISWI bound to the nucleosome with long linker DNA. The dotted line illustrates NegC under stressed condition. (f) Proposed model of ISWI bound to the nucleosome with short linker DNA, in which the Brace helix is partly disrupted and NegC binds to core2 intramolecularly. The HSS domain is omitted.
原文链接
http://www.nature.com/nature/journal/vaop/ncurrent/full/nature20590.html