近期,清华大学医学院/北京生物结构前沿研究中心李海涛课题组在Cell Insight杂志在线发表了题为“Molecular basis for protein histidine N1-specific methylation of the “His-x-His” motifs by METTL9”的封面论文。该研究利用METTL9的底物之一,内质网膜上的锌离子转运蛋白SLC39A7的“HxH”基序为底物,通过综合运用生物化学与结构生物学技术,揭示了METTL9实现“HxH”基序特异性识别的分子基础,并提出了METTL9特异性催化后一位“H”的N1甲基化的催化方式。该研究还发现METTL9催化的底物组氨酸甲基化与锌离子识别之间存在竞争性关系,并提出METTL9对蛋白底物中存在的串联“HxH”基序具有“从C到N”的连续性甲基化倾向。与研究较为充分的赖氨酸和精氨酸甲基化相比,组氨酸甲基化的生理功能和调控正成为一个新兴领域。该工作所揭示出的充满特异性的METTL9催化机制预示着组氨酸甲基化研究的光明未来(Specify the Bright Future of Hme)。
蛋白质翻译后修饰通过引起蛋白质的结构和理化性质的改变,在生命活动和细胞命运决定中发挥着至关重要的作用。而作为最主要的翻译后修饰之一,蛋白质的甲基化修饰通常是由甲基转移酶 (methyltransferases, MTases) 催化,将S-腺苷甲硫氨酸 (S-adenosylmethionine, SAM, 也称为AdoMet) 上的活性甲基基团直接转移到诸如赖氨酸、精氨酸、组氨酸、谷氨酸、谷氨酰胺和半胱氨酸等氨基酸侧链上[1]。
表观遗传学领域内有大量研究已经证明了赖氨酸和精氨酸甲基化对于生物调节和细胞响应等诸多功能非常重要。而最近的研究则逐渐揭示了组氨酸甲基化在各种生物途径中发挥着重要作用[2]。高灵敏度蛋白质组学研究鉴定出了多个哺乳动物组织和细胞的蛋白质组中存在数百个组氨酸甲基化位点[3, 4]。同时随着多个组氨酸甲基转移酶如SETD3、CARNMT1以及METTL18的发现和研究[5-13],组氨酸甲基化对于生命活动的重要性也引起了越来越多研究者的关注和重视。
组氨酸侧链的咪唑环可以分别发生N1或者N3位置的甲基化,并由此导致组氨酸的物理化学性质发生改变,如分子体积和疏水性的增加,组氨酸质子化 (protonation)状态和互变异构状态(tautomerization)的固定[14]。因此细胞利用组氨酸甲基化所带来的理化性质改变作为通过改变蛋白质性质从而影响蛋白质功能的一种策略,可以对于包括二价金属离子(Zn2+, Cu2+, Fe2+)结合[15],以组氨酸为核心的酶促反应,蛋白质复合物组装[10]以及蛋白质翻译控制[13]等大量生命活动产生调控作用。
2021年,Pål Ø. Falnes 课题组和Wen Pan课题组分别报道了一个全新的组氨酸N1甲基转移酶,METTL9,可以催化多个哺乳动物细胞和组织中约80%的组氨酸N1甲基化[15, 16]。同时还发现了METTL9对于“Histidine-x-Histidine”基序(以下简称为“HxH”,其中x为小侧链氨基酸)的识别倾向性,但是关于METTL9如何实现其对于组氨酸N1甲基化的催化的特异性以及对于“HxH”基序的识别倾向性的分子机制尚不明确,同时,为何METTL9可以负责催化哺乳动物细胞中约80%的组氨酸N1甲基化的原因也不清楚。
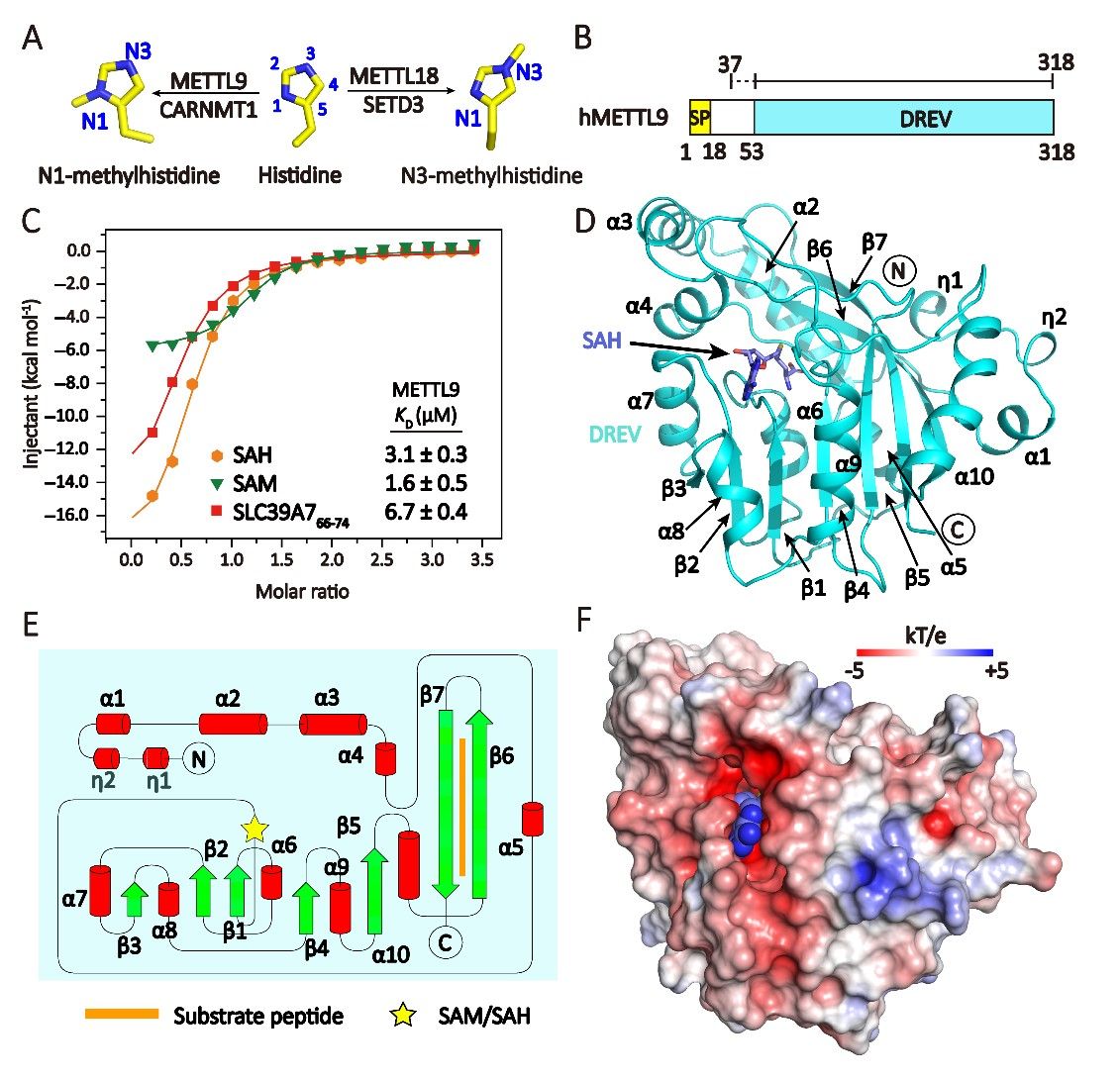
图1. METTL9整体结构
为了揭示METTL9识别和催化的分子机制,首先运用晶体学手段,解析了METTL9蛋白和辅因子SAH的结构,通过结构对比,发现METTL9的催化核心结构域:DREV结构域 (DORA reverse strand domain)符合传统的7BS甲基转移酶家族(seven beta-strand methyltransferase family)催化结构域的核心特征,即催化结构域由7个β片层和夹在它们中间的6个α螺旋组成(图1)。同时METTL9通过一个狭小的负电口袋实现了对于辅因子SAH的识别,多对氢键和疏水作用结合在一起,完成了对于SAH的腺苷环和甲硫氨酸骨架的联合识别,随后对于SAH识别口袋附近的METTL9突变体与SAH的等温滴定量热 (isothermal titration calorimetry, ITC) 结合实验也证明了这些氨基酸对于SAH识别发挥的关键作用(图2)。
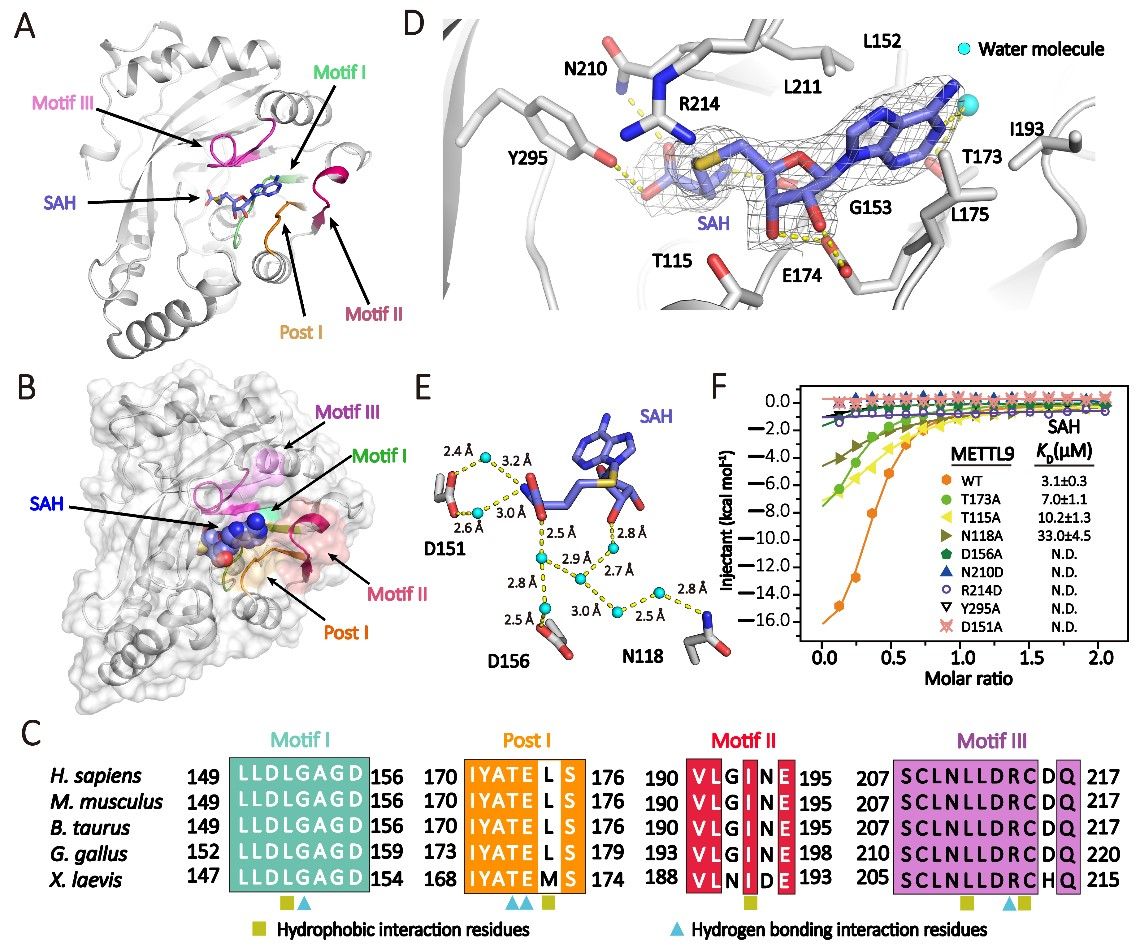
图2. METTL9对辅因子SAH的识别
随后研究者们又选择了一个内质网和高尔基体上非常重要的锌离子转运蛋白,SLC39A7,同时也是METTL9的底物,进行了METTL9底物识别和催化的分子机制的研究。首先研究者们解析了METTL9和SAH以及SLC39A765-70多肽的复合物结构。结构信息显示,底物多肽被深深的包埋进入了带负电的METTL9的底物结合口袋中,底物的溶剂可接触面积 (solvent accessible surface area, ASA) 高达72%,证明了METTL9即使只通过对于核心的“HxH”基序的识别,就可以具有较高的底物结合能力,这也就解释了METTL9可以催化大量底物的原因(图3)。
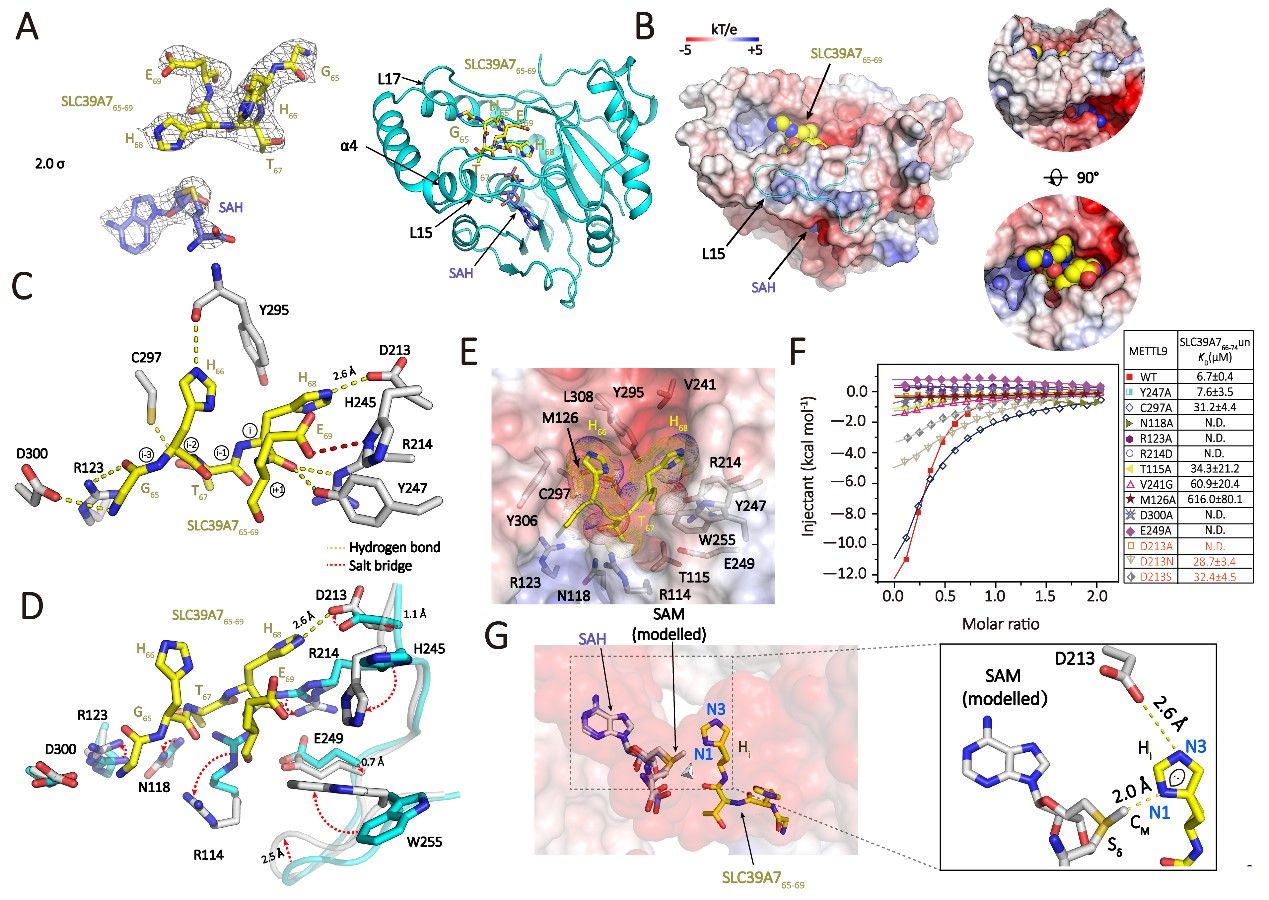
图3. METTL9对底物的识别
对于底物识别的细节分析显示,“HxH”基序中的两个组氨酸都被极度适配的包裹在识别口袋中,两个识别口袋均以疏水氨基酸为主,这些疏水氨基酸既提供了疏水互作以增强对于底物组氨酸的识别同时又通过空间体积的限制,阻止了其他氨基酸的结合。ITC结合实验和基于LC-MS的METTL9甲基化酶活实验也证明了这些结构信息,同时发现METTL9除了可以识别并催化“HxH”基序中的后一个组氨酸“Hi”,同时也会特异性识别前一个组氨酸“Hi-2”,“Hi-2”必须是未甲基化的组氨酸才能发生“Hi”的甲基化,替换成其他氨基酸或甲基化的组氨酸均几乎没有酶活,因此得出了METTL9通过“Hi-2”的特异性识别来引导“Hi”催化的特异性机制(图4)。
“HxH”基序中的“x”也对于底物的识别发挥了作用,结构信息显示,“x”插入了一个空间体积极小的口袋中,由于组成口袋的疏水氨基酸的空间位阻的限制,大侧链氨基酸由于分子体积过大,无法插入口袋,只有如“ANGST”这类小侧链氨基酸才能被口袋识别,即METTL9通过以空间位阻为主的互作方式实现了对于 “x”的选择性。
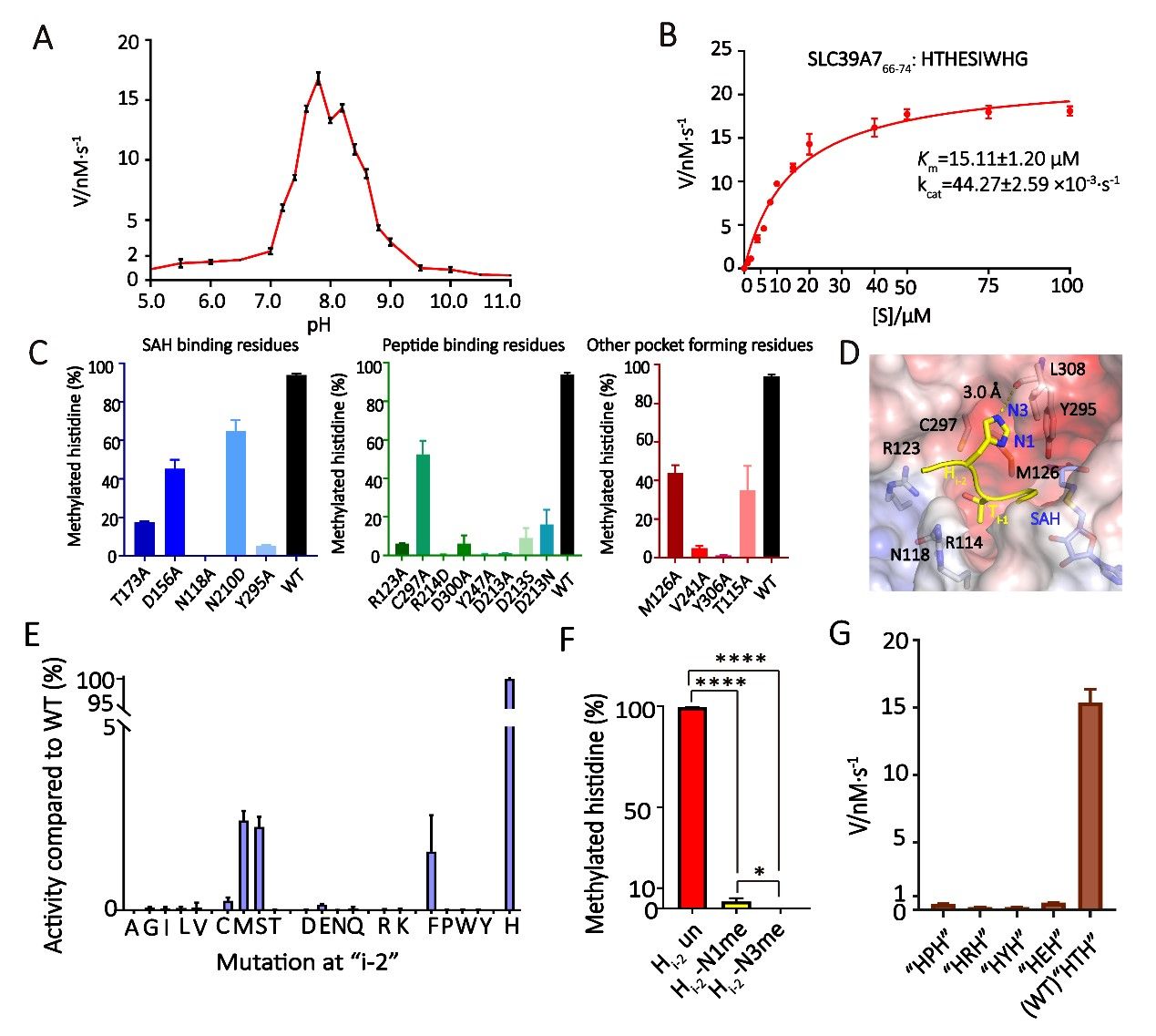
图4. METTL9催化特性的研究
研究者们同时还发现,METTL9的D213可以和被甲基化的组氨酸“Hi”的N3位置形成氢键互作。底物的插入会导致METTL9的D213侧链发生构象调整以与“Hi”的N3形成更强的氢键互作。而根据SN2甲基化反应的机制,这一氢键互作可以使N1位置处于去质子化 (deprotonated) 状态,从而使其可以被SAM上的活性甲基基团可以直接亲核攻击N1,完成N1的甲基加成反应。并且将D213替换成无法形成氢键的丙氨酸后,METTL9与底物的结合和催化能力被完全打破,而替换成可以形成弱氢键的丝氨酸或天冬酰胺后,结合和催化能力减弱,证明了D213与N3的氢键互作对于N1特异性甲基化的作用。
在细胞中,具有“HxH”基序的蛋白质经常作为如锌离子、亚铁离子和铜离子等二价金属离子的螯合蛋白存在的。为了研究METTL9催化的组氨酸甲基化是否会影响底物蛋白质对于锌离子的结合,研究者们首先以多个串联的“HxH”基序为底物进行了METTL9的酶活实验,结果发现锌离子的存在会显著性抑制METTL9的甲基化酶活,而EDTA-Na的处理会使酶活得到恢复。同时ITC结合实验还证明,随着反应时间的增加和甲基化水平的上升,底物对于锌离子的结合能力会逐渐减弱,证明了METTL9催化的组氨酸甲基化和锌离子结合之间存在竞争关系,这对于进一步深入理解和发掘组氨酸甲基化的功能提供了重要的线索(图5)。
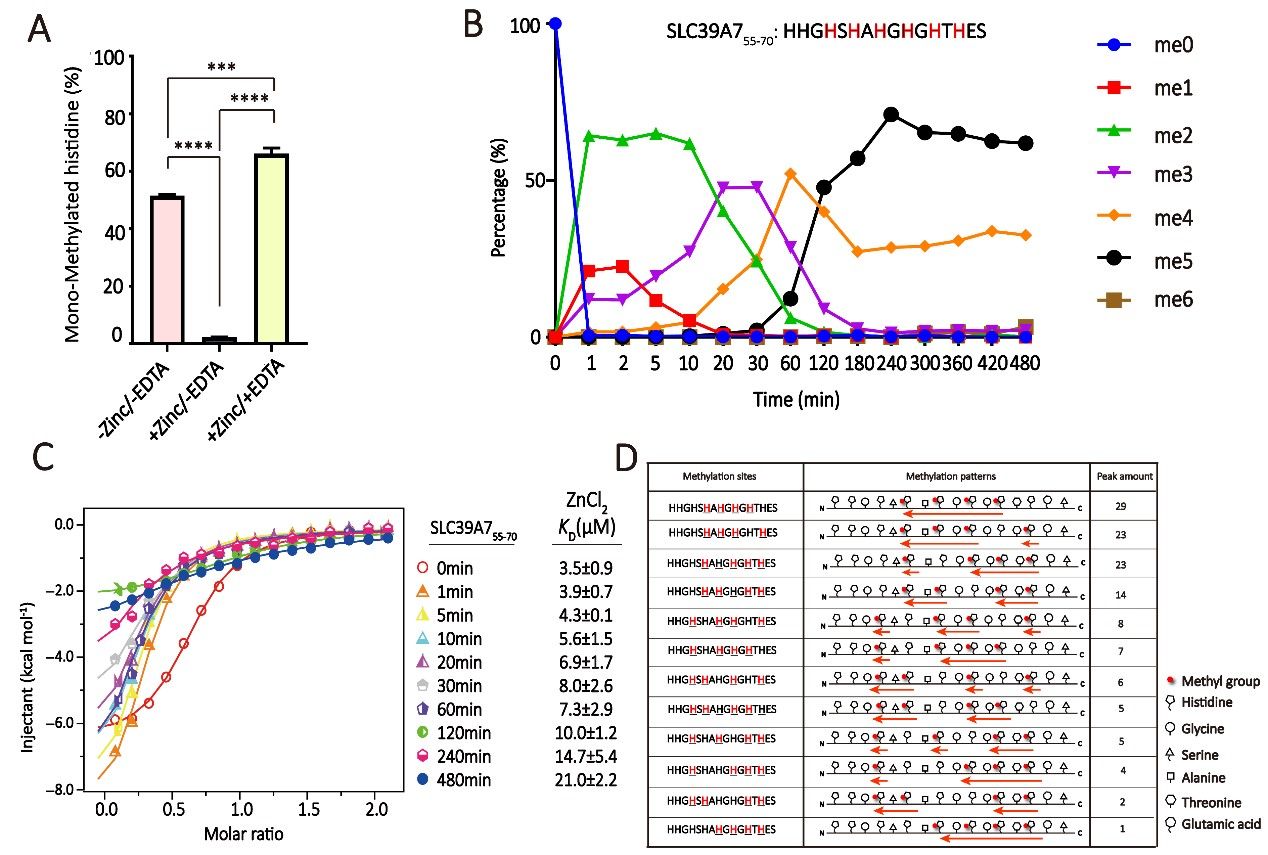
图5. METTL9催化的底物组氨酸甲基化与锌离子结合的竞争关系
此外,研究者还通过LC-MS/MS的手段对于串联“HxH”基序的甲基化状态进行了时序性检测。他们发现,METTL9在催化串联“HxH”基序时会倾向于从C端向N端进行连续甲基化。从一方面而言,“Hi-2”的甲基化会抑制“Hi”的甲基化,因此导致从N端向C端的甲基化无法发生,从另一方面而言,虽然由于METTL9在反应起始时可以随机结合在串联“HxH”基序的不同位置导致甲基化起始状态不同,但是由于未甲基化的“Hi-2”会引导“Hi”的甲基化而“Hi”的甲基化会导致“Hi”脱离催化位点从而使邻近的“Hi-2”插入从而开始下一轮甲基化反应,所以METTL9会呈现出对于连续性的C端向N端甲基化的倾向性。正是由于这一倾向性的存在,在细胞环境下,串联“HxH”基序的高甲基化是很难发生的,因为METTL9起始甲基化的位置是随机的,这也体现了对于METTL9调控底物功能的限制。
与目前已经报道过的组氨酸甲基转移酶如SETD3, CARNMT1以及METTL18相比,METTL9具有识别和催化上的一系列特异性。通过结构对比和分析我们发现,METTL9在底物识别模式上与另一个组氨酸N1甲基转移酶CARNMT1相似,两者都是通过底物组氨酸咪唑环的两端的疏水氨基酸通过疏水互作固定组氨酸方向,随后通过与N3的氢键固定N3,实现N1特异性甲基化。但是与METTL9相比,CARNMT1倾向于自身二聚化,导致了其底物识别口袋被封闭,同时两者的底物识别口袋方向并不一致,从而导致了CARNMT1只能识别短肽底物如二肽的carnosine而METTL9可以识别蛋白质底物(图6)。而N3甲基转移酶SETD3则不同,它通过一个特异性的“头对头”的方式完成底物识别,并且咪唑环并未完全固定,在识别时会发生偏转,但是同样也通过与N1的氢键互作完成了对于N3的特异性催化。
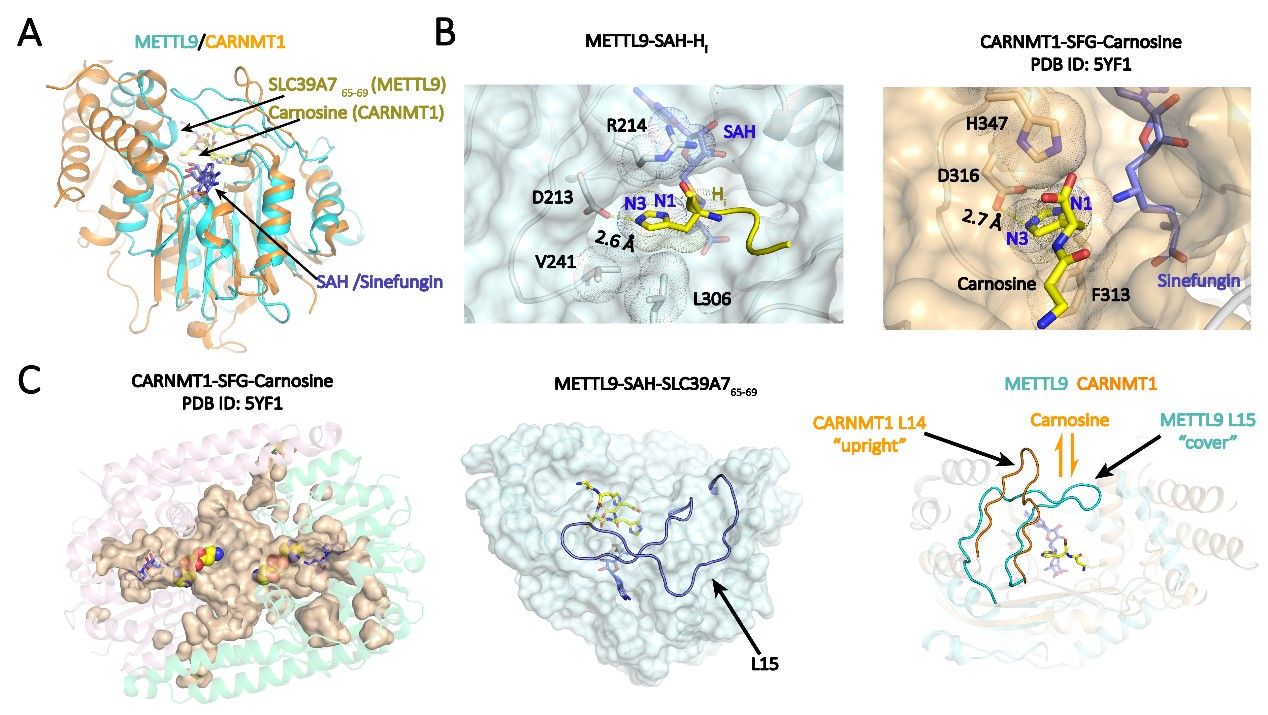
图6. 不同组氨酸甲基转移酶之间的结构对比
清华大学医学院博士生赵文涛、周洋为论文共同第一作者。清华大学博士生李采怿、毕于聪参与了本研究工作,中国科学院大连化学物理研究所叶明亮教授、王科云博士在质谱数据分析方面提供了专业指导。清华大学医学院/北京生物结构前沿研究中心李海涛教授为通讯作者。本项目得到自然科学基金委重大研究计划集成项目、科技部国家重点研发计划资助。衍射数据收集得到上海同步辐射光源提供大力支持。