2018年7月16日,清华大学生命科学学院、结构生物学高精尖创新中心陈春来课题组在学术期刊《核酸研究》(Nucleic Acids Research)杂志在线发表了题为《mRNA下游假结体的构象可塑性对核糖体的动态构象和移位速率的调制》(Translocation kinetics and structural dynamics of ribosomes are modulated by the conformational plasticity of downstream pseudoknots)的研究论文。本工作利用单分子荧光共振能量转移技术(single-molecule FRET)揭示出下游二级结构主要阻碍核糖体移位过程后期的反应速率,且二级结构的构象可塑性与其对移位过程速率的影响和诱导程序核糖体移码的能力有很强的相关性。综合以上结果,本工作揭示了mRNA二级结构的构象可塑性对核糖体翻译和程序核糖体移码的调控机制。
在翻译过程中,mRNA的二级结构能够阻碍核糖体在mRNA上的移动,并是诱导核糖体发生-1位的程序性移码的必要元件之一。早期研究表明二级结构诱导核糖体发生-1位的程序性移码的能力与其机械稳定性和热稳定性无关,而与其构象可塑性显著相关。然而,研究者并不清楚二级结构的构象可塑性是如何影响翻译过程中核糖体的动态构象变化和翻译速率。
单分子荧光共振能量转移技术(single-molecule FRET)是检测生物大分子构象变化的有利工具。本研究通过对tRNA和核糖体蛋白进行位点特异性的荧光标记,利用基于全内反射荧光显微镜的单分子FRET手段实时灵敏地捕捉单个核糖体颗粒在翻译过程中的动态构象变化和翻译速率,揭示了构象可塑性高的mRNA二级结构主要在核糖体与二级结构相接触后的第一个翻译循环里显著地影响核糖体自身构象变化和移位过程的后期反应速率。基于实验发现,提出以下分子模型(示意图)。核糖体在mRNA上的移位反应分为动力冲程(power-stroke)机制主导的前期过程和热棘轮(thermal-ratcheting)机制主导的后期过程(示意图中黑色箭头和曲线)。在动力冲程(power-stroke)过程后,由于核糖体与mRNA的相对移动,将打开二级结构的1-2个碱基对,破坏其稳定结构。而构象可塑性强的mRNA二级结构会在其稳定结构被破坏后,快速转化形成一个新的稳定结构。这将降低移位过程中间态(INT)的自由能,提高后期热棘轮(thermal-ratcheting)步骤的能垒,减缓移位过程后期的反应速率(示意图中蓝色箭头和曲线),并在移码序列存在时诱导核糖体的-1位程序移码。
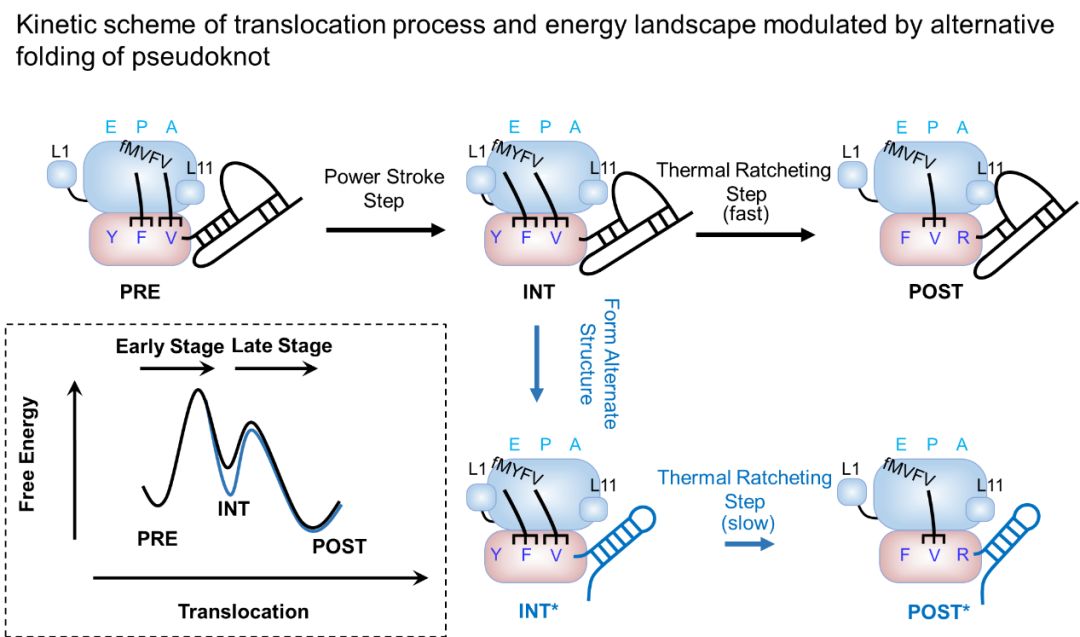
示意图: mRNA二级结构的构象可塑性调控核糖体转位过程的动力学模型
清华大学生命科学学院陈春来研究员和宾大医学院Yale E. Goldman教授为本文共同通讯作者。清华大学生命学院CLS项目2015级博士生吴博为本文的第一作者,生命学院2017级博士生孙瑞瑞和2015级博士生彭思佳参与了本工作。本工作获得了北京结构生物学高精尖创新中心、清华-北大生命科学联合中心及国家自然科学基金委的经费支持。
原文链接:








